第五章 模型后处理
作者:Trent Hauck
译者:飞龙
协议:CC BY-NC-SA 4.0
5.1 K-fold 交叉验证 这个秘籍中,我们会创建交叉验证,它可能是最重要的模型后处理验证练习。我们会在这个秘籍中讨论 k-fold 交叉验证。有几种交叉验证的种类,每个都有不同的随机化模式。K-fold 可能是一种最熟知的随机化模式。
准备 我们会创建一些数据集,之后在不同的在不同的折叠上面训练分类器。值得注意的是,如果你可以保留一部分数据,那是最好的。例如,我们拥有N = 1000的数据集,如果我们保留 200 个数据点,之后使用其他 800 个数据点之间的交叉验证,来判断最佳参数。
工作原理 首先,我们会创建一些伪造数据,之后测试参数,最后,我们会看看结果数据集的大小。
1 2 3 4 >>> N = 1000 >>> holdout = 200 >>> from sklearn.datasets import make_regression >>> X, y = make_regression(1000 , shuffle=True )
既然我们拥有了数据,让我们保留 200 个点,之后处理折叠模式。
1 2 3 >>> X_h, y_h = X[:holdout], y[:holdout] >>> X_t, y_t = X[holdout:], y[holdout:]>>> from sklearn.cross_validation import KFold
K-fold 给了我们一些选项,来选择我们想要多少个折叠,是否让值为下标或者布尔值,是否打算打乱数据集,最后是随机状态(主要出于再现性)。下标实际上会在之后的版本中溢出。假设它为True。
让我们创建交叉验证对象:
1 >>> kfold = KFold(len (y_t), n_folds=4 )
现在,我们可以迭代 k-fold 对象:
1 2 3 4 5 6 7 8 9 >>> output_string = "Fold: {}, N_train: {}, N_test: {}" >>> for i, (train, test) in enumerate (kfold): print output_string.format (i, len (y_t[train]), len (y_t[test])) Fold: 0 , N_train: 600 , N_test: 200 Fold: 1 , N_train: 600 , N_test: 200 Fold: 2 , N_train: 600 , N_test: 200 Fold: 3 , N_train: 600 , N_test: 200
每个迭代都应该返回相同的分割大小。
工作原理 可能很清楚,但是 k-fold 的原理是迭代折叠,并保留 1/n_folds * N个数据,其中N是我们的len(y_t)。
从 Python 的角度看,交叉验证对象拥有一个迭代器,可以通过in运算符来访问。通常,对于编写交叉验证对象的包装器来说比较实用,它会迭代数据的子集。例如我们可能拥有一个数据集,它拥有数据点的重复度量,或者我们可能拥有一个病人的数据集,每个病人都拥有度量。
我们打算将它们组合起来,并对其使用 Pandas。
1 2 3 4 5 6 7 >>> import numpy as np >>> import pandas as pd>>> patients = np.repeat(np.arange(0 , 100 , dtype=np.int8), 8 )>>> measurements = pd.DataFrame({'patient_id' : patients, 'ys' : np.random.normal(0 , 1 , 800 )})
既然我们拥有了数据,我们仅仅打算保留特定的顾客,而不是数据点。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 >>> custids = np.unique(measurements.patient_id) >>> customer_kfold = KFold(custids.size, n_folds=4) >>> output_string = "Fold: {}, N_train: {}, N_test: {}" >>> for i, (train, test) in enumerate(customer_kfold): train_cust_ids = custids[train] training = measurements[measurements.patient_id.isin( train_cust_ids)] testing = measurements[~measurements.patient_id.isin( train_cust_ids)] print output_string.format(i, len(training), len(testing)) Fold: 0, N_train: 600, N_test: 200 Fold: 1, N_train: 600, N_test: 200 Fold: 2, N_train: 600, N_test: 200 Fold: 3, N_train: 600, N_test: 200
5.2 自动化交叉验证 我们会查看如何使用 Sklearn 自带的交叉验证,但是我们也可以使用一个辅助函数,来自动化执行交叉验证。这类似于 Sklearn 中其它对象,如何被辅助函数和流水线包装。
准备 首先,我们需要创建样例分类器,它可以是任何东西,决策树、随机森林,以及其他。对我们来说,它是随机森林。我们之后会创建数据集,并使用交叉验证函数。
工作原理 首先导入ensemble 模块来开始:
1 2 >>> from sklearn import ensemble >>> rf = ensemble.RandomForestRegressor(max_features='auto' )
好的,所以现在,让我们创建一些回归数据:
1 2 >>> from sklearn import datasets >>> X, y = datasets.make_regression(10000 , 10 )
既然我们拥有了数据,我们可以导入cross_validation模块,并获取我们将要使用的函数:
1 2 3 4 5 6 >>> from sklearn import cross_validation>>> scores = cross_validation.cross_val_score(rf, X, y)>>> print scores[ 0.86823874 0.86763225 0.86986129 ]
工作原理 很大程度上,它会委托给交叉验证对象。一个不错的事情是,函数会并行处理交叉验证。
我们可开启详细模式:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 >>> scores = cross_validation.cross_val_score(rf, X, y, verbose=3 , cv=4 ) [CV] no parameters to be set [CV] no parameters to be set , score=0.872866 - 0.7 s [CV] no parameters to be set [CV] no parameters to be set , score=0.873679 - 0.6 s [CV] no parameters to be set [CV] no parameters to be set , score=0.878018 - 0.7 s [CV] no parameters to be set [CV] no parameters to be set , score=0.871598 - 0.6 s [Parallel(n_jobs=1 )]: Done 1 jobs | elapsed: 0.7 s [Parallel(n_jobs=1 )]: Done 4 out of 4 | elapsed: 2.6 s finished
我们可以看到,在每次迭代中,我们都调用函数来获得得分。我们也知道了模型如何运行。
同样值得了解是的,我们可以对我们尝试拟合的模型,获取预测得分。我们也会讨论如何创建你自己的评分函数。
5.3 使用 ShuffleSplit 交叉验证 ShuffleSplit是最简单的交叉验证技巧之一。这个交叉验证技巧只是将数据的样本用于指定的迭代数量。
准备 ShuffleSplit是另一个简单的交叉验证技巧。我们会指定数据集中的总元素,并且它会考虑剩余部分。我们会浏览一个例子,估计单变量数据集的均值。这有点类似于重采样,但是它说明了一个原因,为什么我们在展示交叉验证的时候使用交叉验证。
操作步骤 首先,我们需要创建数据集。我们使用 NumPy 来创建数据集,其中我们知道底层的均值。我们会对半个数据集采样,来估计均值,并看看它和底层的均值有多接近。
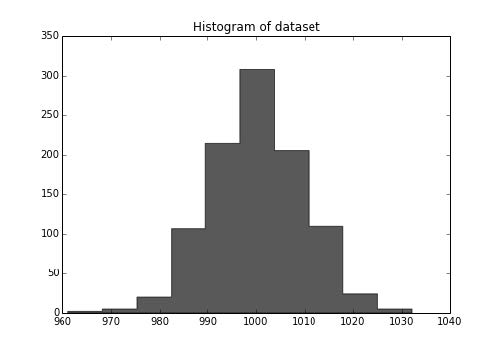
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 >>> import numpy as np>>> true_loc = 1000 >>> true_scale = 10 >>> N = 1000 >>> dataset = np.random.normal(true_loc, true_scale, N)>>> import matplotlib.pyplot as plt>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.hist(dataset, color='k' , alpha=.65 , histtype='stepfilled' ); >>> ax.set_title("Histogram of dataset" );>>> f.savefig("978-1-78398-948-5_06_06.png" )
NumPy 输出如下:
现在,让我们截取前一半数据集,并猜测均值:
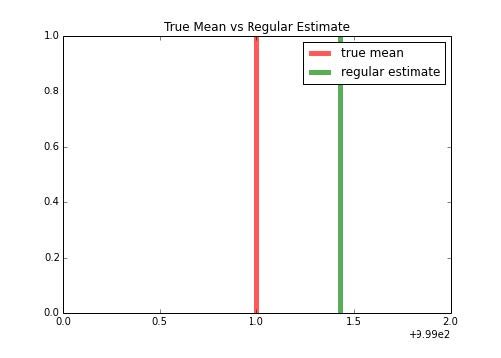
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 >>> from sklearn import cross_validation>>> holdout_set = dataset[:500 ] >>> fitting_set = dataset[500 :]>>> estimate = fitting_set[:N/2 ].mean()>>> import matplotlib.pyplot as plt>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.set_title("True Mean vs Regular Estimate" )>>> ax.vlines(true_loc, 0 , 1 , color='r' , linestyles='-' , lw=5 , alpha=.65 , label='true mean' ) >>> ax.vlines(estimate, 0 , 1 , color='g' , linestyles='-' , lw=5 , alpha=.65 , label='regular estimate' ) >>> ax.set_xlim(999 , 1001 )>>> ax.legend()>>> f.savefig("978-1-78398-948-5_06_07.png" )
输出如下:
现在,我们可以使用ShuffleSplit 在多个相似的数据集上拟合估计值。
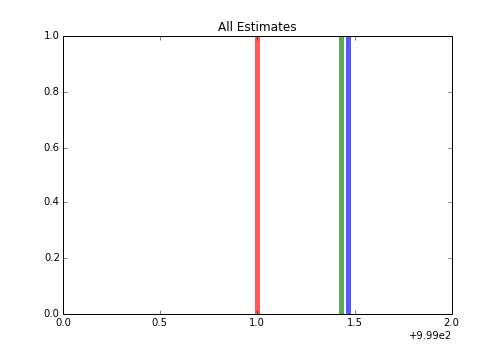
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 >>> from sklearn.cross_validation import ShuffleSplit>>> shuffle_split = ShuffleSplit(len (fitting_set))>>> mean_p = []>>> for train, _ in shuffle_split: mean_p.append(fitting_set[train].mean()) shuf_estimate = np.mean(mean_p) >>> import matplotlib.pyplot as plt>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.vlines(true_loc, 0 , 1 , color='r' , linestyles='-' , lw=5 , alpha=.65 , label='true mean' ) >>> ax.vlines(estimate, 0 , 1 , color='g' , linestyles='-' , lw=5 , alpha=.65 , label='regular estimate' ) >>> ax.vlines(shuf_estimate, 0 , 1 , color='b' , linestyles='-' , lw=5 , alpha=.65 , label='shufflesplit estimate' ) >>> ax.set_title("All Estimates" ) >>> ax.set_xlim(999 , 1001 )>>> ax.legend(loc=3 )
输出如下:
我们可以看到,我们得到了类似于预期的估计值,但是我们可能使用多个样本来获取该值。
5.4 分层的 k-fold 这个秘籍中,我们会快速查看分层的 k-fold 估值。我们会浏览不同的秘籍,其中分类的表示在某种程度上是不平衡的。分层的 k-fold 非常不错,因为他的模式特地为维持分类的比例而设计。
准备 我们打算创建一个小型的数据集。这个数据集中,我们随后会使用分层的 k-fold 验证。我们想让它尽可能小,以便我们查看变化。对于更大的样本,可能并不是特别好。
我们之后会绘制每一步的分类比例,来展示如何维护分类比例。
1 2 3 4 >>> from sklearn import datasets >>> X, y = datasets.make_classification(n_samples=int (1e3 ), weights=[1. /11 ])
让我们检查分类的总体权重分布:
1 2 >>> y.mean()0.90300000000000002
90.5% 的样本都是 1,其余为 0。
操作步骤 让我们创建分层 k-fold 对象,并通过每个折叠来迭代。我们会度量为 1 的verse 比例。之后,我们会通过分割数字来绘制分类比例,来看看是否以及如何发生变化。这个代码展示了为什么它非常好。我们也会对基本的ShuffleSplit绘制这个代码。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 >>> from sklearn import cross_validation>>> n_folds = 50 >>> strat_kfold = cross_validation.StratifiedKFold(y, n_folds=n_folds) >>> shuff_split = cross_validation.ShuffleSplit(n=len (y), n_iter=n_folds) >>> kfold_y_props = [] >>> shuff_y_props = []>>> for (k_train, k_test), (s_train, s_test) in zip (strat_kfold, >>> shuff_split): kfold_y_props.append(y[k_train].mean()) shuff_y_props.append(y[s_train].mean())
现在,让我们绘制每个折叠上的比例:
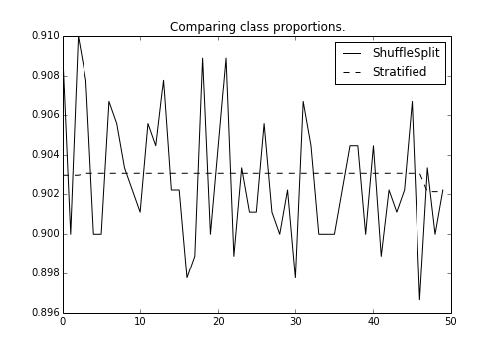
1 2 3 4 5 6 7 8 9 10 11 12 13 >>> import matplotlib.pyplot as plt>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.plot(range (n_folds), shuff_y_props, label="ShuffleSplit" , color='k' ) >>> ax.plot(range (n_folds), kfold_y_props, label="Stratified" , color='k' , ls='--' ) >>> ax.set_title("Comparing class proportions." )>>> ax.legend(loc='best' )
输出如下:
我们可以看到,分层的 k-fold 的每个折叠的比例,在每个折叠之间是稳定的。
工作原理 分层 k-fold 的原理是选取y值。首先,获取所有分类的比例,之后将训练集和测试集按比例划分。这可以推广到多个标签:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 >>> import numpy as np>>> three_classes = np.random.choice([1 ,2 ,3 ], p=[.1 , .4 , .5 ], size=1000 ) >>> import itertools as it>>> for train, test in cross_validation.StratifiedKFold(three_classes, 5 ): print np.bincount(three_classes[train]) [ 0 90 314 395 ] [ 0 90 314 395 ] [ 0 90 314 395 ] [ 0 91 315 395 ] [ 0 91 315 396 ]
我们可以看到,我们得到了每个分类的样例大小,正好是训练集合测试集的比例。
5.5 菜鸟的网格搜索 这个秘籍中,我们打算使用 Python 来介绍基本的网格搜索,并且使用 Sklearn 来处理模型,以及 Matplotlib 来可视化。
准备 这个秘籍中,我们会执行下面这些东西:
同样,我们训练的模型是个基本的决策树分类器。我们的参数空间是 2 维的,有助于我们可视化。
1 2 criteria = {gini, entropy} max_features = {auto, log2, None}
参数空间是criteria和max_features的笛卡尔积。
我们会了解如何使用itertools来迭代这个空间。
让我们创建数据集来开始:
1 2 >>> from sklearn import datasets >>> X, y = datasets.make_classification(n_samples=2000 , n_features=10 )
操作步骤 之前我们说,我们使用网格搜索来调整两个参数 – criteria和max_features``criteria和max_features。我们需要将其表示为 Python 集合,之后使用itertools.product来迭代它们。
不错,所以既然我们拥有了参数空间,让我们迭代它并检查每个模型的准确率,它们由参数指定。之后,我们保存这个准确率,便于比较不同的参数空间。我们也会使用以50, 50划分的测试和训练集。
1 2 3 4 5 6 7 8 9 10 11 12 import numpy as np train_set = np.random.choice([True , False ], size=len (y)) from sklearn.tree import DecisionTreeClassifier accuracies = {} for criterion, max_feature in parameter_space: dt = DecisionTreeClassifier(criterion=criterion, max_features=max_feature) dt.fit(X[train_set], y[train_set]) accuracies[(criterion, max_feature)] = (dt.predict(X[~train_set]) == y[~train_set]).mean() >>> accuracies {('entropy' , None ): 0.974609375 , ('entropy' , 'auto' ): 0.9736328125 , ('entropy' , 'log2' ): 0.962890625 , ('gini' , None ): 0.9677734375 , ('gini' , 'auto' ): 0.9638671875 , ('gini' , 'log2' ): 0.96875 }
所以现在我们拥有了准确率和它的表现。让我们可视化它的表现。
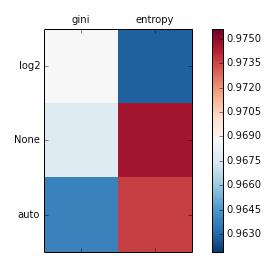
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 >>> from matplotlib import pyplot as plt >>> from matplotlib import cm >>> cmap = cm.RdBu_r >>> f, ax = plt.subplots(figsize=(7 , 4 )) >>> ax.set_xticklabels(['' ] + list (criteria)) >>> ax.set_yticklabels(['' ] + list (max_features)) >>> plot_array = [] >>> for max_feature in max_features: m = [] >>> for criterion in criteria: m.append(accuracies[(criterion, max_feature)]) plot_array.append(m) >>> colors = ax.matshow(plot_array, vmin=np.min (accuracies.values()) - 0.001 , vmax=np.max (accuracies.values()) + 0.001 , cmap=cmap) >>> f.colorbar(colors)
输出如下:
很容易看到哪个表现最好。单元你可以使用爆破方式看到它如何进一步处理。
工作原理 原理很简单,我们只需要执行下列步骤:
选取一系列参数
迭代它们并求得每一步的准确率
通过可视化来寻找最佳的表现
5.6 爆破网格搜索 这个秘籍中,我们会使用 Sklearn 做一个详细的网格搜索。这基本和上一章的事情相同,但是我们使用内建方法。
我们也会浏览一个执行随机化优化的示例。这是个用于爆破搜索的替代方案。本质上,我们花费一些计算周期,来确保搜索了整个空间。我们在上一个秘籍中比较冷静,但是,你可以想想拥有多个步骤的模型,首先对缺失数据进行估算,之后使用 PCA 降低维度来分类。你的参数空间可能非常大,非常块,因此,搜索一部分空间是有利的。
准备 我们需要下列步骤来开始:
创建一些数据集
之后创建LogisticRegression 对象,训练我们的模型
之后,我们创建搜索对象,GridSearch 和RandomizedSearchCV
工作原理 执行下列代码来创建一些分类数据
1 2 >>> from sklearn.datasets import make_classification>>> X, y = make_classification(1000 , n_features=5 )
现在,我们创建逻辑回归对象:
1 2 >>> from sklearn.linear_model import LogisticRegression>>> lr = LogisticRegression(class_weight='auto' )
我们需要指定打算搜索的参数。对于GridSearch,我们可以指定所关心的范围,但是对于RandomizedSearchCV,我们实际上需要指定相同空间上的分布:
1 2 3 4 5 6 7 8 >>> lr.fit(X, y)LogisticRegression(C=1.0 , class_weight={0 : 0.25 , 1 : 0.75 }, dual=False , fit_intercept=True , intercept_scaling=1 , penalty='l2' , random_state=None , tol=0.0001 ) >>> grid_search_params = {'penalty' : ['l1' , 'l2' ], 'C' : [1 , 2 , 3 , 4 ]}
我们需要做的唯一一个修改,就是将C参数描述为概率分布。我们现在使其保持简单,虽然我们使用scipy来描述这个分布。
1 2 3 4 >>> import scipy.stats as st >>> import numpy as np>>> random_search_params = {'penalty' : ['l1' , 'l2' ], 'C' : st.randint(1 , 4 )}
工作原理 现在,我们要训练分类器了。原理是将lr作为参数传给搜索对象。
1 2 >>> from sklearn.grid_search import GridSearchCV, RandomizedSearchCV>>> gs = GridSearchCV(lr, grid_search_params)
GridSearchCV 实现了和其他方法相同的 API:
1 2 3 4 5 6 7 8 9 >>> gs.fit(X, y)GridSearchCV(cv=None , estimator=LogisticRegression(C=1.0 , class_weight='auto' , dual=False , fit_intercept=True , intercept_scaling=1 , penalty='l2' , random_state=None , tol=0.0001 ), fit_params={}, iid=True , loss_func=None , n_jobs=1 , param_grid={'penalty' : ['l1' , 'l2' ], 'C' : [1 , 2 , 3 , 4 ]}, pre_dispatch='2*n_jobs' , refit=True , score_func=None , scoring=None , verbose=0 )
我们可以看到, param_grid参数中的penalty 和C都是数组。
为了评估得分,我们可以使用网格搜索的grid_scores_ 属性。我们也打算寻找参数的最优集合。我们也可以查看网格搜索的边际表现。
1 2 3 4 5 6 7 8 9 >>> gs.grid_scores_[mean: 0.90300 , std: 0.01192 , params: {'penalty' : 'l1' , 'C' : 1 }, mean: 0.90100 , std: 0.01258 , params: {'penalty' : 'l2' , 'C' : 1 }, mean: 0.90200 , std: 0.01117 , params: {'penalty' : 'l1' , 'C' : 2 }, mean: 0.90100 , std: 0.01258 , params: {'penalty' : 'l2' , 'C' : 2 }, mean: 0.90200 , std: 0.01117 , params: {'penalty' : 'l1' , 'C' : 3 }, mean: 0.90100 , std: 0.01258 , params: {'penalty' : 'l2' , 'C' : 3 }, mean: 0.90100 , std: 0.01258 , params: {'penalty' : 'l1' , 'C' : 4 }, mean: 0.90100 , std: 0.01258 , params: {'penalty' : 'l2' , 'C' : 4 }]
我们可能打算获取最大得分:
1 2 3 4 5 >>> gs.grid_scores_[1 ][1 ]0.90100000000000002 >>> max (gs.grid_scores_, key=lambda x: x[1 ])mean: 0.90300 , std: 0.01192 , params: {'penalty' : 'l1' , 'C' : 1 }
获取的参数就是我们的逻辑回归的最佳选择。
5.7 使用伪造的估计器来比较结果 这个秘籍关于创建伪造的估计其。这并不是一个漂亮或有趣的东西,但是我们值得为最后构建的模型创建一个参照点。
准备 这个秘籍中,我们会执行下列任务:
创建一些随机数据
训练多种伪造的估计器
我们会对回归数据和分类数据来执行这两个步骤。
操作步骤 首先,我们创建随机数据:
1 2 3 4 5 6 7 8 9 >>> X, y = make_regression()>>> from sklearn import dummy>>> dumdum = dummy.DummyRegressor()>>> dumdum.fit(X, y)DummyRegressor(constant=None , strategy='mean' )
通常,估计器仅仅使用数据的均值来做预测。
1 2 3 >>> dumdum.predict(X)[:5 ]array([ 2.23297907 , 2.23297907 , 2.23297907 , 2.23297907 , 2.23297907 ])
我们可以尝试另外两种策略。我们可以提供常数来做预测(就是上面命令中的constant=None),也可以使用中位值来预测。
如果策略是constant,才会使用提供的常数。
让我们看一看:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 >>> predictors = [("mean" , None ), ("median" , None ), ("constant" , 10 )] >>> for strategy, constant in predictors: dumdum = dummy.DummyRegressor(strategy=strategy, constant=constant) >>> dumdum.fit(X, y) >>> print "strategy: {}" .format (strategy), "," .join(map (str , dumdum.predict(X)[:5 ])) strategy: mean 2.23297906733 ,2.23297906733 ,2.23297906733 ,2.23297906733 ,2 .23297906733 strategy: median 20.38535248 ,20.38535248 ,20.38535248 ,20.38535248 ,20.38535 248 strategy: constant 10.0 ,10.0 ,10.0 ,10.0 ,10.0
我们实际上有四种分类器的选项。这些策略类似于连续情况,但是适用于分类问题:
1 2 3 4 >>> predictors = [("constant" , 0 ), ("stratified" , None ), ("uniform" , None ), ("most_frequent" , None )]
我们也需要创建一些分类数据:
1 2 3 4 5 6 7 8 9 10 11 >>> X, y = make_classification()>>> for strategy, constant in predictors: dumdum = dummy.DummyClassifier(strategy=strategy, constant=constant) dumdum.fit(X, y) print "strategy: {}" .format (strategy), "," .join(map (str , dumdum.predict(X)[:5 ])) strategy: constant 0 ,0 ,0 ,0 ,0 strategy: stratified 1 ,0 ,0 ,1 ,0 strategy: uniform 0 ,0 ,0 ,1 ,1 strategy: most_frequent 1 ,1 ,1 ,1 ,1
工作原理 最好在最简单的模型上测试你的模型,这就是伪造的估计器的作用。例如,在一个模型中,5% 的数据是伪造的。所以,我们可能能够训练出一个漂亮的模型,而不需要猜测任何伪造。
我们可以通过使用分层(stratified)策略来床架买模型,使用下面的命令。我们也可以获取一个不错的示例,关于为什么分类的不均等会导致问题:
1 2 3 4 5 6 7 8 9 10 11 12 >>> X, y = make_classification(20000 , weights=[.95 , .05 ])>>> dumdum = dummy.DummyClassifier(strategy='most_frequent' )>>> dumdum.fit(X, y)DummyClassifier(constant=None , random_state=None , strategy='most_ frequent' ) >>> from sklearn.metrics import accuracy_score>>> print accuracy_score(y, dumdum.predict(X))0.94575
我们实际上经常是正确的,但关键不是这个。关键是,这就是我们的基线。如果我们不能为伪造数据创建模型,并且比这个更准确,它就不值得我们花时间。
5.8 回归模型评估 我们已经学过了如何量化分类中的误差,现在我们讨论连续问题中的误差。例如,我们尝试预测年龄而不是性别。
准备 像分类一样,我们伪造一些数据,之后绘制变化。我们开始会很简单,之后逐步变复杂。数据是模拟的线性模型。
1 2 3 m = 2 b = 1 y = lambda x: m * x + b
同时,导入我们的模块:
1 2 3 >>> import numpy as np >>> import matplotlib.pyplot as plt >>> from sklearn import metrics
操作步骤 我们会执行下列操作:
使用y来生成y_actual
使用y_actual加上一些err生成y_prediction'
绘制差异
遍历不同的度量并绘制它们
让我们同时关注步骤 1 和 2,并且创建一个函数来帮助我们。这与我们刚刚看的相同,但是我们添加一些功能来指定误差(如果是个常量则为偏差)。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 >>> def data (x, m=2 , b=1 , e=None , s=10 ): """ Args: x: The x value m: Slope b: Intercept e: Error, optional, True will give random error """ if e is None : e_i = 0 elif e is True : e_i = np.random.normal(0 , s, len (xs)) else : e_i = e return x * m + b + e_i
既然我们已经拥有了函数,让我们定义y_hat和y_actual。我们会以便利的方法来实现:
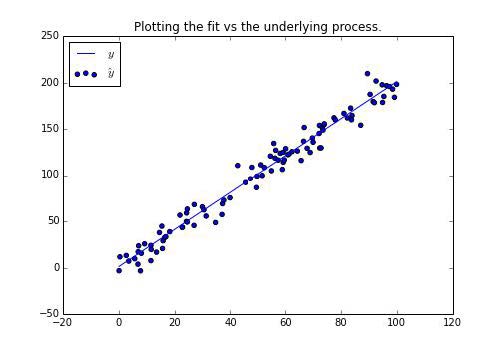
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 >>> from functools import partial>>> N = 100 >>> xs = np.sort(np.random.rand(N)*100 )>>> y_pred_gen = partial(data, x=xs, e=True ) >>> y_true_gen = partial(data, x=xs)>>> y_pred = y_pred_gen() >>> y_true = y_true_gen()>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.set_title("Plotting the fit vs the underlying process." ) >>> ax.scatter(xs, y_pred, label=r'$\hat{y}$' )>>> ax.plot(xs, y_true, label=r'$y$' )>>> ax.legend(loc='best' )
输出如下:
仅仅为了验证输出,我们要计算经典的残差。
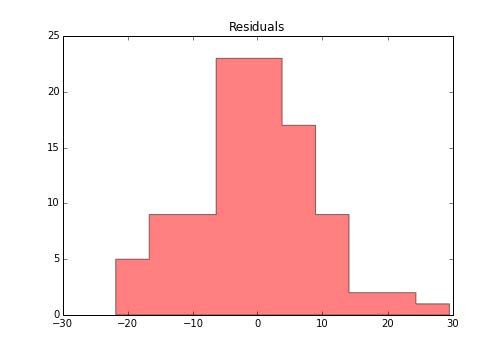
1 2 3 4 5 6 >>> e_hat = y_pred - y_true>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.set_title("Residuals" ) >>> ax.hist(e_hat, color='r' , alpha=.5 , histtype='stepfilled' )
输出如下:
看起来不错。
工作原理 现在让我们看看度量。
首先,一种度量就是均方误差。
1 MSE(y_trus, y_pred) = E((y_trus - y_pred)^2)
1 mse = ((y_trus - y_pred) ** 2 ).mean()
你可以使用下面的代码来计算均方误差值:
1 2 3 >>> metrics.mean_squared_error(y_true, y_pred)93.342352628475368
要注意,这个代码会惩罚更大误差。要注意,我们这里所做的是,将模型的可能的损失函数应用于测试数据。
另一个萱萱个就是平均绝对差。我们需要计算差异的绝对值。如果我们不这么做,我们的值就可能接近于零,也就是分布的均值:
1 MAD(y_trus, y_pred) = E(|y_trus - y_pred|)
1 mad = np.abs (y_trus - y_pred).mean()
最终的选项是 R 平方,它是 1 减去拟合模型的均方误差,与整体均值的军方误差的比值。随着比值接近于 0,R 平方接近于 1。
1 rsq = 1 - ((y_trus - y_pred) ** 2 ).sum () / ((y_trus - y_trus.mean()) ** 2 ).sum ()
1 2 3 >>> metrics.r2_score(y_true, y_pred)0.9729312117010761
R 平方是描述性的,它不提供模型准确性的清晰感觉。
5.9 特征选取 这个秘籍以及后面那个都关于自动特征选取。我喜欢将其看做参数调整的特征替换。就像我们做交叉验证来寻找合适的通用参数,我们可以寻找合适的特征通用子集。这涉及到几种不同方式。
最简单的想法就是到那边了选取。其它方法涉及到处理特征的组合。
特征选取的一个额外好处就是,它可以减轻数据收集的负担。想象你已经在一个很小的数据子集上构建了模型。如果一切都很好,你可能打算扩展来预测数据的整个子集。如果是这样,你可以减少数据收集的工作量。
准备 在单变量选取中,评分函数又出现了。这次,它们会定义比较度量,我们可以用它来去掉一些特征。
这个秘籍中,我们会训练带有 10000 个特征的回归模型,但是只有 1000 个点。我们会浏览多种单变量特征选取方式。
1 2 >>> from sklearn import datasets >>> X, y = datasets.make_regression(1000 , 10000 )
既然我们拥有了数据,我们会使用多种方式来比较特征。当你进行文本分析,或者一些生物信息学分析时,这是个非常常见的情况。
操作步骤 首先,我们需要导入feature_selection模块。
1 2 >>> from sklearn import feature_selection >>> f, p = feature_selection.f_regression(X, y)
这里,f就是和每个线性模型的特征之一相关的 f 分数。我们之后可以比较这些特征,并基于这个比较,我们可以筛选特征。p是f值对应的 p 值。
在统计学中,p值是一个值的概率,它比检验统计量的当前值更极端。这里f值检验统计量。
1 2 3 4 5 >>> f[:5 ] array([ 1.06271357e-03 , 2.91136869e+00 , 1.01886922e+00 , 2.22483130e+00 , 4.67624756e-01 ]) >>> p[:5 ] array([ 0.97400066 , 0.08826831 , 0.31303204 , 0.1361235 , 0.49424067 ])
我们可以看到,许多p值都太大了。我们更想让p值变小。所以我们可以将 NumPy 从工具箱中取出来,并且选取小于.05的p值。这些就是我们用于分析的特征。
1 2 3 4 5 >>> import numpy as np >>> idx = np.arange(0 , X.shape[1 ]) >>> features_to_keep = idx[p < .05 ] >>> len (features_to_keep)501
你可以看到,我们实际上保留了相当大的特征总量。取决于模型的上下文,我们可以减少p至。这会减少保留的特征数量。
另一个选择是使用VarianceThreshold 对象。我们已经了解一些了。但是重要的是理解,我们训练模型的能力,基本上是基于特征所产生的变化。如果没有变化,我们的特征就不能描述独立变量的变化。根据文档,良好的特征可以用于非监督案例,因为它并不是结果变量。
我们需要设置起始值来筛选特征。为此,我们选取并提供特征方差的中位值。
1 2 3 4 5 6 >>> var_threshold = feature_selection.VarianceThreshold(np.median(np. var(X, axis=1 ))) >>> var_threshold.fit_transform(X).shape(1000 , 4835 )
我们可以看到,我们筛选了几乎一半的特征,或多或少就是我们的预期。
工作原理 通常,所有这些方式的原理都是使用单个特征来训练基本的模型。取决于它是分类问题还是回归问题,我们可以使用合适的评分函数。
让我们观察一个更小的问题,并可视化特征选取如何筛选特定的特征。我们使用第一个示例的相同评分函数,但是仅仅有 20 个特征。
1 2 3 >>> X, y = datasets.make_regression(10000 , 20 )>>> f, p = feature_selection.f_regression(X, y)
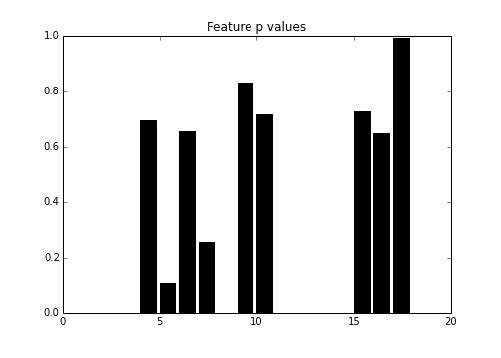
现在,让我们绘制特征的p值,我们可以看到筛选和保留哪个特征:
1 2 3 4 5 6 >>> from matplotlib import pyplot as plt>>> f, ax = plt.subplots(figsize=(7 , 5 ))>>> ax.bar(np.arange(20 ), p, color='k' ) >>> ax.set_title("Feature p values" )
输出如下:
我们可以看到,许多特征没有保留,但是保留了一些特征。
5.10 L1 范数上的特征选取 我们打算实现一些相似的理念,我们在套索回归的秘籍中见过他们。在那个米几种,我们查看了含有 0 系数的特征数量。
现在我们打算更进一步,并使用 L1 范数来预处理特征。
准备 我们要使用糖尿病数据集来拟合回归。首先,我们要使用ShuffleSplit 交叉验证来训练基本的LinearRegression 模型,之后,我们使用LassoRegression 来寻找 L1 惩罚为 0 的系数。我们希望它能帮助我们避免过拟合,也就是说这个模型非常特定于所训练的数据。换句话说,如果过拟合的话,模型并不能推广到外围的数据。
我们打算执行下列步骤:
加载数据集
训练基本的线性回归模型
使用特征选取来移除不提供信息的特征
重新训练线性回归,并与特征完整的模型相比,它拟合得多好。
操作步骤 首先加载数据集:
1 2 >>> import sklearn.datasets as ds >>> diabetes = ds.load_diabetes()
让我们导入度量模块的mean_squared_error函数,以及cross_validation模块的 ShuffleSplit交叉验证函数。
1 2 3 4 >>> from sklearn import metrics >>> from sklearn import cross_validation>>> shuff = cross_validation.ShuffleSplit(diabetes.target.size
现在训练模型,我们会跟踪ShuffleSplit每次迭代中的均方误差。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 >>> mses = [] >>> for train, test in shuff: train_X = diabetes.data[train] train_y = diabetes.target[train] test_X = diabetes.data[~train] test_y = diabetes.target[~train] lr.fit(train_X, train_y) mses.append(metrics.mean_squared_error(test_y, lr.predict(test_X))) >>> np.mean(mses)2856.366626198198
所以既然我们做了常规拟合,让我们在筛选系数为 0 的特征之后再检查它。让我们训练套索回归:
1 2 3 4 5 6 7 8 9 10 >>> from sklearn import feature_selection >>> from sklearn import cross_validation>>> cv = linear_model.LassoCV() >>> cv.fit(diabetes.data, diabetes.target) >>> cv.coef_array([ -0. , -226.2375274 , 526.85738059 , 314.44026013 , -196.92164002 , 1.48742026 , -151.78054083 , 106.52846989 , 530.58541123 , 64.50588257 ])
我们会移除第一个特征。我使用 NumPy 数组来表示模块中包含的列。
1 2 3 >>> import numpy as np >>> columns = np.arange(diabetes.data.shape[1 ])[cv.coef_ != 0 ] >>> columns array([1 , 2 , 3 4 , 5 , 6 , 7 , 8 , 9 ])
好的,所以现在我们使用特定的特征来训练模型(请见下面代码中的列):
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 >>> l1mses = []>>> for train, test in shuff: train_X = diabetes.data[train][:, columns] train_y = diabetes.target[train] test_X = diabetes.data[~train][:, columns] test_y = diabetes.target[~train] lr.fit(train_X, train_y) l1mses.append(metrics.mean_squared_error(test_y, lr.predict(test_X))) >>> np.mean(l1mses) 2861.0763924492171 >>> np.mean(l1mses) - np.mean(mses) 4.7097662510191185
我们可以看到,即使我们移除了不提供信息的特征,模型依然不怎么样。这种情况不是始终发生。下一部分中,我们会比较模型间的拟合,其中有很多不提供信息的特征。
工作原理 首先,我们打算创建回归数据集,带有很多不提供信息的特征:
1 >>> X, y = ds.make_regression(noise=5 )
让我们训练普通的回归:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 >>> mses = []>>> shuff = cross_validation.ShuffleSplit(y.size)>>> for train, test in shuff: train_X = X[train] train_y = y[train] test_X = X[~train] test_y = y[~train] lr.fit(train_X, train_y) mses.append(metrics.mean_squared_error(test_y, lr.predict(test_X))) >>> np.mean(mses)879.75447864034209
现在我们可以以相同个过程来使用套索回归:
1 2 3 4 5 6 >>> cv.fit(X, y)LassoCV(alphas=None , copy_X=True , cv=None , eps=0.001 , fit_intercept=True , max_iter=1000 , n_alphas=100 , n_jobs=1 , normalize=False , positive=False , precompute='auto' , tol=0.0001 , verbose=False )
我们会再次创建列。这是个很好的模式,让我们能够制定要包含的列。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 >>> import numpy as np >>> columns = np.arange(X.shape[1 ])[cv.coef_ != 0 ] >>> columns[:5 ] array([11 , 15 , 17 , 20 , 21 ,])>>> mses = []>>> shuff = cross_validation.ShuffleSplit(y.size)>>> for train, test in shuff: train_X = X[train][:, columns] train_y = y[train] test_X = X[~train][:, columns] test_y = y[~train] lr.fit(train_X, train_y) mses.append(metrics.mean_squared_error(test_y, lr.predict(test_X))) >>> np.mean(mses)15.755403220117708
我们可以看到,我们在模型的训练中获得了极大的提升。这刚好解释了,我们需要承认,不是所有特征都需要或者应该放进模型中。
5.11 使用 joblib 保存模型 这个秘籍中,我们打算展示如何保存模型,便于以后使用。例如,你可能打算实际使用模型来预测结果,并自动做出决策。
准备 这个秘籍中,我们会执行下列任务:
训练我们要保存的模型
导入 joblib 并保存模型
操作步骤 为了使用 joblib 保存我们的模型,可以使用下面的代码:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 >>> from sklearn import datasets, tree>>> X, y = datasets.make_classification()>>> dt = tree.DecisionTreeClassifier() >>> dt.fit(X, y)DecisionTreeClassifier(compute_importances=None , criterion='gini' , max_depth=None , max_features=None , max_leaf_nodes=None , min_density=None , min_samples_leaf=1 , min_samples_split=2 , random_state=None , splitter='best' ) >>> from sklearn.externals import joblib>>> joblib.dump(dt, "dtree.clf" )['dtree.clf' , 'dtree.clf_01.npy' , 'dtree.clf_02.npy' , 'dtree.clf_03.npy' , 'dtree.clf_04.npy' ]
工作原理 上面的下面的原理是,保存对象状态,可以重新加载进 Sklearn 对象。要注意,对于不同的模型类型,模型的状态拥有不同的复杂度级别。
出于简单的因素,将我们要保存的东西看做一种方式,我们提供出入来预测结果。对于回归来说很简单,简单的线性代数就足以。但是,对于像是随机森林的模型,我们可能拥有很多颗树。这些树可能拥有不同的复杂度级别,比较困难。
更多 我们可以简单随机森林模型的大小:
1 2 3 4 5 6 7 8 9 10 11 12 >>> from sklearn import ensemble>>> rf = ensemble.RandomForestClassifier() >>> rf.fit(X, y)RandomForestClassifier(bootstrap=True , compute_importances=None , criterion='gini' , max_depth=None , max_features='auto' , max_leaf_nodes=None , min_density=None , min_samples_leaf=1 , min_samples_split=2 , n_estimators=10 , n_jobs=1 , oob_score=False , random_state=None , verbose=0 )
我打算省略输出,但是总之,我的机器上一共有 52 个输出文件。
1 2 3 4 5 6 7 8 9 >>> joblib.dump(rf, "rf.clf" ) ['rf.clf' , 'rf.clf_01.npy' , 'rf.clf_02.npy' , 'rf.clf_03.npy' , 'rf.clf_04.npy' , 'rf.clf_05.npy' , 'rf.clf_06.npy' , ... ]